デザイン & 開発理論 2
生物学の画像はここ 15 年位で飛躍的に複雑化してきている。例えば、1995 年位までの論文ではアナログフィルムからそのまま持ってきた画像も多々見かけたが、これ以降はフォトショップなどで画質調整したデジタル画像が主流となっている。また同じデジタル画像でもこれまでは2次元であったものが、最近では3次元・4次元情報を含む画像も多く見受けられるようになった。
3次元画像作成において、z-スライス中から最も明るいシグナルをピックアップして画像を生成する Maximal Intensity Projection(MIP)法は、その単純なアルゴリズム故に問題が少ない。ただ、シグナルが3次元的に込み入った構造では全てが、ベッタリと塗りつぶされて飽和した絵として表現されてしまう。
そうした複雑な構造を観察するには direct volume rendering(DVR)法にて3次元構造を反映した絵を作成する必要がある。ただ、ここで問題なのが3次元画像を生成するのに、「ソフトウェア中で何をやっているのか分からない。なぜか知らないが連続スライスで見えているシグナルが、立体再構成をすると消えてしまう。」といったことが現存するほぼ全ての3次元再構成ソフトウェアで起こることである。また全ての再構成ソフトウェアで、内部処理が生物学者に対して明らかにされていない。
「自分が使っているツールが何をやっているのか分からない。だけど結果が得られる。」基礎分野の科学者にとってこれは由々しき事態であるといっても良いと思う。
私自身、多くの三次元再構成ソフトウェアを使用・解析し、二年半に渡って FluoRender というソフトウェアをプログラマの Yong Wan と共に作成してきて最近分かったことがある。「この世の中にある自分が試した限りの全てのソフトウェア*1 は、画像生成をいい加減に行っている。また生物学者のワークフローにフィットさせるようなソフトウェアデザインを行っていない。」
*1. 参考までこれまで私が試したソフトウェアのいくつかを述べる。
volocity5, Imaris6, amira5, V3D, voxx, imagesurfer, Osirix, daime, Eikona3D, MRIcro, volume viewer in ImageJ, 3D slicer, bioview3D, Seg3D など
もし、共焦点データから三次元画像をリアルタイムで真剣に最高の品質で生成しようとすると、2010 年のPCハードウェアの性能では最高レベルのゲーミンググラフィックカードがどうしても必要になる。このハードウェア制限無しで共焦点データから画像をリアルタイムで生成しているソフトウェアは全て、なんらかのシグナル情報を切り落として画像を生成している。シグナル情報を切り落とさなくても、データの解像度をソフトウェア内部で落として(小さいテクスチャとしてレンダリングして)、それを拡大リサイズしてディスプレイに表示したり。なども常套手段として見受けられる。
また、ほとんどのフリーウェアは VTK や ITK といった三次元再構成ライブラリを使用しているため、画質に制限がある。こういったライブラリは汎用性を重視しているため、どうしてもスピードやカスタマイズ性に問題があり、結果として画像のクオリティーを追求することができない。ただこれら汎用ライブラリを使用するプログラマの側からすると、たったの2行のコードでボリュームレンダリング機能を自分のソフトウェアに追加できるのだから便利なことこの上ない。
しかし FluoRender ではオリジナルソースコードにてレンダリングライブラリ・コアを主に Yong が書いている*2 。それ故に詳細な画質調整が可能であり、反面、結果としてこの部分のチューニングに膨大な時間がかかる*3 。だがこうした努力によって、存在するシグナルを切り落とさずに全ての情報を表示可能なソフトウェアとして仕上けることができた。またオリジナルソースコード故に Composite / Layer モードを Yong は比較的簡単に搭載することができた。
*2. オリジナルソースコードを用いることで、生じるデメリットもある。これまでに何度か他の研究所のプログラマーと FluoRender の共同開発の話が立ち上がった。しかし汎用ライブラリを用いていないために Plugins の追加などが簡単に行なえず、全ての話が流れてしまった。しかしながらユーザーのことを考えると、品質の悪いレンダリングで多機能のものよりも、現在の高画質でファイル読み込みが速い方がソフトウェアとしての価値があると我々は考えている。
*3. 基本的に全ての画質調整パラメーターは線形で推移する。しかし全てのパラメーターの上限と下限値、レンダリングスライスの枚数、陰影強度、透明度の実装法、MIPと陰影のバランス、スームージングフィルターの値など他にも多岐に渡って私の目で画像の良し悪しを判断して調整を行っている。
ただこれにも良し悪しがあり、ソフトウェアを売る側からすると、あれもこれも出来ますと言って売る方が楽であり、顧客もより多くの機能を搭載したソフトウェアの方が自分たちの欲求をより満たしてくれるのではと期待する。なので現状の FluoRender のように「生物学者のワークフローにフィットし、ハイクオリティな3次元・4次元再構成とムービーの作成に特化しています。」と売り込んでも、これから共焦点顕微鏡データの可視化を行いたい研究者にはいまいちソフトウェアの価値が伝わりにくい。(これまでいくつものソフトウェアを試してきた玄人には、幸いにしてその価値を分かってもらえてはいるが。)
だが機能ありきの戦略でソフトウェアを作っていくと、後になっての画質改善は難しいものとなる。その理由は、レンダリングの計算結果を元にして次の画像解析・処理の機能を書いているので、レンダリングの部分を変えてしまうとその後のコードを大幅に改変しないといけなくなるからである。今後 FluoRender にも画像解析の機能を搭載していく予定だが、すでに 2.5 年を visualization のみに費やし、画質に関しては最高に近い品質に仕上がっているので、我々にはこうした問題が起こらないのではないかと考えている。
では、FluoRende の中で何をやっているのか、いくつかの項目について見てみよう。
レンダリングスライス枚数、角度の調整
レンダリングスライスという言葉は多くの生物学者にとって耳慣れないものである。しかしそのスライスを目にしている人は実は非常に多い。例えば3Dレンダリング時にオブジェクトを回したとき、等高線のようなパターンがチラチラとしているのを見たことがあるだろうか?これはレンダリングスライス枚数が少ないためにスライス間のギャップが見えているのである。
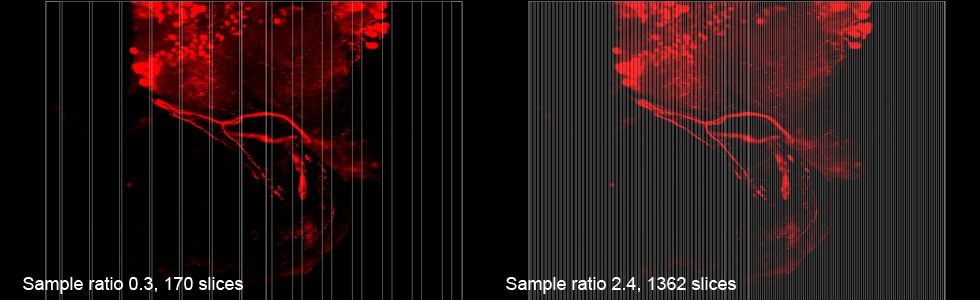
多くの再構成ソフトウェアではこのスライスベースのレンダリングを用いている。しかしながら、スライス枚数の調整機能を持つものは非常に少ない。FluoRender では Sample Rate というパラメーターにてレンダリングスライス枚数の調整を行なっている。以下の写真は、左が Sample Rate 0.3 の時のもので、170枚のレンダリングスライス、右が同 2.4 の時で 1362 枚のレンダリングスライスからなるオブジェクト。白い縦線はレンダリングスライスを可視化したものである。
またこの時 Sample Rate 2.4 では 0 度の正面からオブジェクトを見たときはおよそ 1000 枚、90 度の真横から見たときにはおよそ 1900 枚までスライスが増加する。この増加はスライス間の距離を一定に保っている故におこる。このサンプルは 0 度の正面から見たときには奥行きが浅く、真横から見たときには奥行きが深くなっている。
このようなデータを回転させたときにスライスの増加が起こらないと、オブジェクトの透明化や陰影の不均一が発生してしまう(ちなみに Imaris6 ではこの透明化が見られる、また複数のソフトウェアで回転時に陰影の点滅が見られる)。ただ、FluoRenderではスライス間の距離を一定に保つ数式をモディファイして、スライスの増加曲線を人の目に対して最適化している。ちなみにレンダリングスライスが増加するため、FluoRender では真横からのレンダリングでスピードが落ちることに気づいている方もおられるかと思う。
また FluoRender でレンダリングを行った時、多くの共焦点データで Sample Rate 2.4 の時に、人の目でスライスが見えなくなる。すなわちこれ以上の数値に設定しても、オーバーサンプリングでスピードが落ちるだけで大きなメリットはない。ただ、一枚の画像からなるサンプルでは Sample Rate を 5 、もしくはそれ以上の数値をタイプしないと、レンダリングスライスの縞模様が見えることとなる。三次元オブジェクトではスライス間でシグナルが混じり合うため、レンダリングスライスのギャップが見えにくい。しかし一枚の画像では混じり合うシグナルがないため、ダイレクトにレンダリングスライスが見えてしまう。
この他にも FluoRender の中ではレンダリングスライスが常にカメラ(画面)に対して垂直になるように計算をしている。これにより、回転時にオブジェクトが透明化するのをさらに抑制している。またレンダリングスライス間での積分には GPU コードを用いており、Yong 自身は書いていない。ちなみにもちろん、こうしたコーディングは全て Yong が行った。私は画質のチェックや円錐形や四角形などのサンプルデータの作成を当時担当した。ただ今こうして画質のチェックと一言でいえるが、当時は最適化するまで 2 ヶ月を要した。
透明度(alpha)、明るいバックグラウンドについて
3次元レンダリングされた生物学データを人が見て、自然だと感じるには何が必要なのか?それはその人が前もって見たオリジナルデータ(連続スライス)に近い画像である必要がある。そのためには全てのボクセルを元の値通りに忠実にレンダリングすればよいのであり、一見なんの問題も無いように見える。
しかしながら問題は別のところにあり、ダイレクトボリュームレンダリングされた絵では、薄暗いシグナルが奥にある明るいシグナルを覆い隠してしまう。これを日常生活に例えると、暗闇の中にある光源を見ることができないということになる。
ではどうして我々はそのような光源を見ることができるのかというと、空気や水が透明だからである。多くのボリュームレンダリングソフトウェア同様、FluoRender でもこの透明度の設定ができる。ここで注意しておきたいのが、3Dでの透明度設定は2Dイメージで行ったのとは結果が異なることである。
2Dイメージで透明度を下げると、明るい部分も暗い部分も等しく透明化される。しかし3Dデータでは三次元でシグナルの加算現象が起こる。レンダリングスライス一枚一枚の中では確かに明るいシグナルも暗いシグナルも同時に透明度が落ちているのだが、明るいシグナルは3次元構造内でより加算される。
結果、3次元レンダリングで透明度を下げると、明るいオブジェクトが暗いオブジェクトに対して優勢になり、我々はその絵を自然だと感じる。
また MIP からの出力は2Dイメージであるために、透明度を下げると明るいシグナルも暗いシグナルも同時に暗くなってしまい、不自然な絵になる。
FluoRender の開発段階で MIP を ON/OFF して画像を比較する場合、MIP ON のたびにいちいちこの透明度を最大値に設定し直す必要があった。なので私は MIP 使用時には、ソフトウェア内部で透明度の値を最大非透明値に設定して、かつスライドバーをグレーアウトさせてユーザーが透明度を変化できないようにデザインした。
また、背景を明るい色でレンダリングしたとき、透明度が低いとオブジェクトの色と背景の色が混じり合って忠実な色を表示できなかった。
そこで Yong がとった方法は、まずオブジェクトを画面に表示する前にメモリ上で黒いバックグラウンドで一度レンダリングする。次にシグナルが無い領域のみをユーザーが選択した明るい色のバックグラウンドに置換するというものであった。
これによりオブジェクト自体は正確な色でレンダリングされつつ、かつユーザーは好みのバックグラウンド色を選べるようになっている。
ボクセル間の十字パターンの改善
オブジェクトを0度の正面から見ると、薄暗いシグナルの部分に細かい十字パターンが見える。これは trilinear interpolation の結果、薄暗いボクセル同士の間でシグナルが落ち込んでギャップが生じ、奥にある明るいシグナルが見えていることに起因する。
数々のフィルタを Yong が試した結果、Minimal フィルタが十字パターンの消去に有効であり、かつオブジェクトの解像度を落とさないで済むことが分かった。しかし私がテストした結果、シグナルそのものが縮んでしまっていて神経線維を拡大したときに見られるプレシナプスのふっくらした感じが無くなっていることに気づいた。
そこで Minimal フィルタをグレースケール 40 以下の薄暗いシグナルのみにかけ、かつ Maximal フィルタの強度をシグナル強度に対して3の平方根で変える(暗いグレースケール値により強く)と元のシグナルを保存できつつ、パターンをキャンセルできることを我々は膨大なテストから見出した。
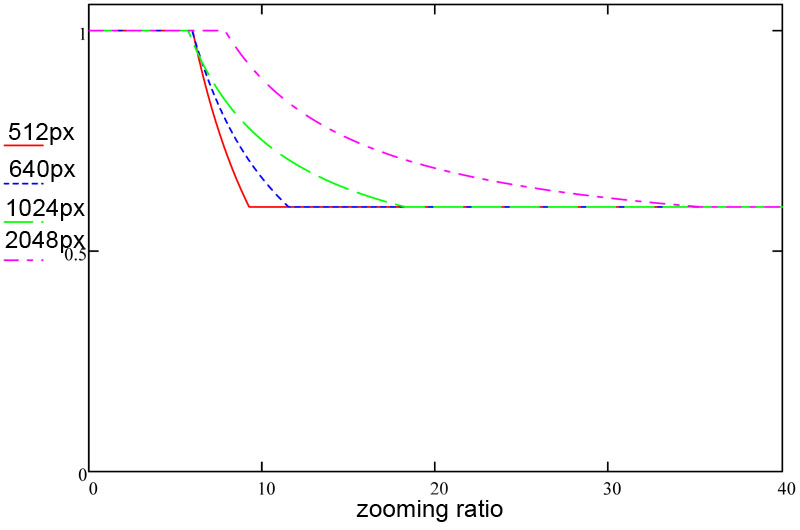
またさらにズーム値ごとの最適なフィルタサイズを 512, 640, 1024, 2048px のデータを用いて私がプロットし、Yong が近似の数式を求め、さらに私がその数式を画像に合わせてファインチューニングするということを行った。結果、ボクセル間の十字パターンをマイルドにしつつ、シグナルの劣化にほとんど影響しないフィルタ強度カーブ(ズーム値に対して)を実装できた。この問題とあとで述べるテクスチャサイズの問題解決に我々は2ヶ月を費やした。
ただ、より自然な結果を求める生物学者の習性を考えると常にフィルタを ON にしておくのは、望ましくないこともあるかと思い、 フィルタの ON/OFF のコントロール項目を加るようにデザインを提案した。
しかしながら私が知る限りの他のソフトウェアではこのような項目は存在せず、ソフトウェア中でどのようなフィルタを用いているのかも明らかにされていない。薄暗いシグナルやスライス一枚のみからなるデータが多くのソフトウェアで表示されないことから、おそらく median フィルタのような強力なノイズキャンセリングフィルタが実装されていると考えられる。
MIP with shading & color map
この機能を搭載しようとしたのには2つの理由がある。1つは生物学者が「自然な感じのレンダリング結果」を好むこと。 MIPの絵は薄暗いシグナルが薄暗く、明るいシグナルは明るく、自然な感じに見える。
またもう一つは伊藤研の後輩である稲垣君(Caltech, Ph.D student)が、立体オブジェクトのグレスケール値をカラーマップで表示する必要に迫られていたこと。共焦点顕微鏡においてシグナルとは、なんらかの遺伝子の発現を示しているため、その発現強度を知りたい研究者は結構多いのではないだろうかと私はなんとなく推測した。
当初、単純にダイレクトボリュームレンダリングにカラーマップをそのまま追加した。しかし明るいシグナルを覆い隠す暗いシグナルの影響で、最大輝度値の表示が出来なかった。そこで MIP を用いて最大輝度を表示するとともに、立体構造の情報も可視化できる方法を我々は模索し始めた。
この方法で問題なのが、MIP は全ての z-スライスから最大輝度のボクセルをもってきて絵を出力している、すなわち MIP からの出力には立体構造情報が含まれていない。
ではどこから立体構造情報をもってくるのか?そこで Yong が考え出した方法は MIP の他にもう一枚、普通のボリュームレンダリングをオブジェクト色を白で行う。次にMIPとの間で「比較暗(フォトショップでいうところの)」をする。すると MIP より暗い陰影のみが取り出せる、というものであった。実際この方法により、MIP でありながら立体構造を表示するということに成功した。
先程のカラーマップの話に戻るが、グレースケールの値をカラーマップで表示する機能は ImageJ にも搭載されており、目新しいものではない。しかしながら私の知る限りでは、画像の明るさを変えるとマップされた色まで変わってしまうソフトウェアしかこれまでに存在していない。
研究者はデータ中のグレースケールの絶対値を知りたいのであって、画像の明るさの値を知りたいのではない。またカラーマップを適用した場合、暗いシグナルを暗く表示する必要はなく、暗いシグナルを明るく見やすく表示できる機能の方が望ましい。
そこで私はカラーマップ機能を FluoRender に追加するときに、マップ中の色をグレースケール値でのみ変えられるようにデザインした。これによりユーザーはカラーマップからの色を保持したまま、画像の明るさを変えられる。この機能は単純で、カラーマップを3次元的にデータに適用し、その出力画像に対して2次元で明るさの調整を行うというものである。
結果、FluoRender に明るさの絶対値を可視化する機能を付加することができた。しかしカラーマップを実装した当初(Ver2.6.1まで)では Yong との間に誤解があり、画像の明るさ調整で色が変わってしまっていた。これまで互いがどこまで理解しているのかを確認する作業を常に行なってきた。だがそれでも大きな誤解が生じていたことがたびたびあり、生物学者とプログラマーの思考方向の差というのがソフトウェア作成において最も大きな壁だと改めて痛感した。
ファイル読み込みのスピードについて、4Dレンダリング
ファイル読み込みスピードの良し悪しは、ソフトウェアを使うユーザーにとって大きな意味を持つ。しかし私が知る限りの再構成ソフトウェアでは、この問題を真剣に解決しようとした形跡がない *4。
*4. 2010 年の IEEE Vis ミーティングでのボリュームレンダリングトークでは、ほとんどがレンダリングスピードの改善、少ないサンプリング時の画質の維持についてであった。しかしこれらは全てレンダリングの前に必要な値を計算する必要があり、こういったアルゴリズムをソフトウェアに搭載すると使用メモリの増大、読み込みスピードの遅延が生じる。しかし大抵の再構成ソフトウェアでは 30MB 程度のファイルの読み込みに 5~10 秒以上かかり、複数のサンプルを読み込むと 1 分以上待たされるなんていうのもザラである。しかし FluoRender では特殊なアルゴリズムを一切搭載せず、必要最低限の計算しかファイル読み込み時に行っていない。それ故に高速なファイル読み込みが可能となっている。ちなみに Yong 曰く、「世の中複雑化することばかりで、シンプルにかつデータローディングスピードを早くするという方向で仕事をしている人はまずいない」
FluoRender をリリースしてから1年間は、レンダリングに必要な gradient magnitude 値をファイル読み込み時にCPUで計算していた。そのため、640px x 640px x 90slices x 3Ch のデータを一般的なPCで読み込むのに 20 秒近くかかっていた。しかし 4D レンダリングを実装しようとしたとき、その読み込み時間が問題となった。
ちなみに Volocity や Imaris といった他のソフトウェアは 3D / 4D 共に、ファイル読み込み時に必要な値を計算してメモリにデータを保存する。そうした場合に問題となるのが、PCのメモリが 12GB しかないけど 4D ファイルが 7GB あって数値計算の結果 13GB もメモリが必要になる、4Dファイルの読み込みに 1 時間以上かかってソフトウェアがクラッシュするなどである。
その問題を解決するために、リアルタイムに 1 time point のみの 4D ファイルをメモリに読み込み→開放するというソフトウェア構造を私はデザインした。4D ファイルの読み込みをリアルタイム化するために Yong と話し合い、gradient magnitude の計算をオンザフライで GPU にて計算させようという方針に決まった。
その機能を実装した結果、レンダリングスピードの落ち込みを 50% に抑えられ、メモリの消費量を 25% まで減らせ、データの読み込みを 20 倍速くできることが分かった。またその時のテストで判明したことに FluoRender ではリアルタイムレンダリングに必要な計算時間のうち 90% を gradient magnitude の値取得・計算に費やしているということがあった。
これはどういうことかというと 32bit レンダリング・ブレンディングや、trilinear interpolation、その他多くのパラメーター調整計算などはほとんどレンダリングのスピードに影響しない。従って現在の一般的な PC の性能では FluoRender の中で行っている 7 つの隣り合うボクセル値の gradient magnitude 値参照・計算が実用限界であるということが分かった。
このリアルタイムファイル読み込みで 4D レンダリングを行う機能を搭載した結果、512px x 385px x 38slices x 2Ch の 4D データを1.5~2 fps(Volocity は同一データを 0.5fps で4Dレンダリング)で、同サイズの 7Ch のデータを 0.5 fpsで 4D レンダリングすることができた。
またユーザーからは 50GB の 4D ファイルのレンダリングに成功したとレポートがあった。その他、使用メモリを劇的に減らせた影響として、私の周りの生物学者の本ソフトウェアの使用法が変わった。これまで数個体分ずつデータを読み込み一つ一つレンダリングしていたものが、一気に 8 個体以上の 3Ch データを読み込んでソフトウェア上でサンプルを比較しながらレンダリングするようになった。
また、極端な例としては 2048px で撮影し、FluoRender 上で 10 倍以上に拡大して目的細胞のみをレンダリングするという使い方まで出てきた。ただ、32bit バージョンのソフトウェアでは通常 1GB または 1.7GB 程度までしかメモリを使用できない制限がある。それ故に 2048px x 2048px x 100slices x 3Ch を読み込むとクラッシュしてしまった。
このため、Ver.2.6 から 64bit バージョンもリリースした。64bit バージョンではグラフィックカードに搭載されたメモリ以上のデータを読み込むと、自動的にシステムメモリにスワップする。その場合はレンダリングが遅くなる、しかしソフトウェアクラッシュすることはなく 2048px x 2048px x 100slices x 7Ch (2.5GB) を GTX280 (1GB) を用いたシステムで読み込むことができた。
高倍率ズームレンダリング時のスピード改善
上記のメモリ使用量の改善の結果、一部のユーザーが巨大なファイルサイズをソフトウェアに読み込み、10 倍以上の高倍率ズームでオブジェクトをレンダリングするようになった。ただそこで問題になるのが、これまでの FluoRender ではズーム倍率を高くすればするほどスピードが遅くなることであった。
この問題を解決するために、我々はテクスチャサイズをズーム倍率に応じて変えるアプローチを用いた。たとえば、1024px のデータを10 倍にズームインしたとき、確かに構造のそのものは見えてはいるが、画面ピクセル単位の詳細な凹凸などは見えない。これはそもそもデータにそこまでの解像度が無いから起こる。
そのような場合、データサイズを縮小してレンダリングし、拡大して結果をディスプレイに表示することでレンダリングスピードを上げることができる(これをテクスチャサイズの縮小という)。このとき実際には解像度が落ちているのだが、解像度の落ち幅をデータの解像度に合わせることで、見かけ上の変化を抑えることができる。
そこで私は 512, 640, 1024, 2048px のデータ群に対して様々なズーム値とテクスチャサイズでレンダリングし、結果をプロットしていった。このときの結果は、画像を劣化させないテクスチャサイズの値をズーム値ごとに求めるというものである。
FluoRender でのズーム値とテクスチャサイズの関係。縦軸はテクスチャのサイズ、横軸はズーム値 (10=1000 in FluoRender)。テクスチャサイズは1から始まり、0.6 まで落ち込む。
テクスチャサイズが1とは画面上の画像サイズとレンダリングしているデータのサイズが同一であることを意味する。最初のテクスチャサイズが1なのは、1から大きくしても小さくしても画素補間によって画質が劣化するからである。
結果、FluoRender では超拡大ズーム時に最大で約3倍のスピードアップをはたし 、かつ実質的な画質劣化を起こすことなくレンダリングすることができるようになった。
2.2 ワークフロー・機能デザインへ進む
|